简介
*问题:*我对R语言中常用的用于质量控制的 "成对 "图有几个问题。 (1)它的空间效率不高,因为它只用了一半的空间来表示数据点;(2)散点图的信息量不大。 当我看这些散点图时,很难说出数据的分布或任何正常化问题。 这对蛋白质组学数据来说尤其如此,因为高动态范围可能会掩盖低丰度点。
*解决方案。*一组MA图(最小-平均)。 MA图显示了一对样品的倍数变化与平均强度。 它可以让你看到样本组之间的差异,我认为这是一个有用的比较单位,可以直观地看到归一化问题。 为了节省空间,我们将不绘制每个样本之间的对比图,而只比较组间样本。
这个方法利用了数据透视的优势,简洁地生成了一个MA图的面板
suppressPackageStartupMessages(library(tidyverse))
library(GGally)
library(DEFormats)
设置数据
我将从模拟数据开始,它类似于基因表达研究。 蛋白质组学的数据集也会类似。 该数据集将有8个样本,其中一半是处理过的,一半是对照。 其中7个样本将大致相同,但样本4与其他样本相比将有3倍的增长,以说明MA图如何帮助识别归一化的问题。
counts <- simulateRnaSeqData(n = 5000, m = 8)
counts[, 4] <- counts[, 4] * 3
targets <- data.frame(sample = colnames(counts), group = c(rep("control", 4), rep("treated", 4)))
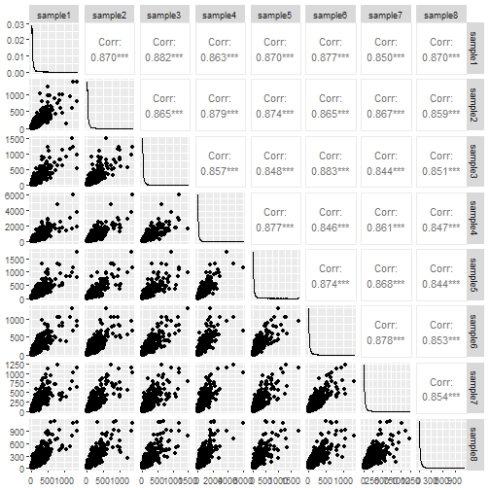
GGally 软件包中的ggpairs 函数在成对图方面做得不错。
ggpairs(data.frame(counts))
成对图告诉我们关于数据的一些情况。 相关性是很好的,如果任何一个样本与其他的样本有很大的不同,就会在散点图中显示出来。 但我不认为它能非常有效地传达信息。
MA图面板
通常情况下,我会先将所有的计数数据透视到一个单列,然后加入元数据。 但是我需要将每个样本的每个基因的对照和处理数据联系起来,所以通常的方法是行不通的。 我花了一些时间才找到解决办法:我们必须分别对对照组和处理组样本进行透视。 因此,对于每个基因,我们将有一个对照样本名称,一个处理样本名称以及对照和处理计数数据。 这些可以用来计算折叠变化和强度。
control_samples <- targets$sample[targets$group == "control"]
treated_samples <- targets$sample[targets$group == "treated"]
data.frame(counts) %>%
rownames_to_column("gene") %>%
pivot_longer(all_of(control_samples), names_to = "control_sample", values_to = "control_count") %>%
pivot_longer(all_of(treated_samples), names_to = "treated_sample", values_to = "treated_count") %>%
mutate(FC = treated_count / control_count) %>%
mutate(Intensity = (treated_count + control_count) / 2)
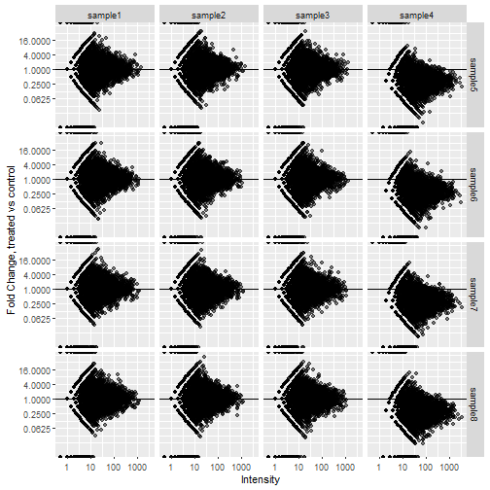
现在要做的就是绘图。 Facet_grid会让我们把样品分割开来。
data.frame(counts) %>%
rownames_to_column("gene") %>%
pivot_longer(all_of(control_samples), names_to = "control_sample", values_to = "control_count") %>%
pivot_longer(all_of(treated_samples), names_to = "treated_sample", values_to = "treated_count") %>%
mutate(FC = treated_count / control_count) %>%
mutate(Intensity = (treated_count + control_count) / 2) %>%
ggplot(aes(x = Intensity, y = FC)) +
geom_point(alpha = 0.5, na.rm = TRUE) +
scale_x_continuous(trans = "log10") +
scale_y_continuous(trans = "log2", breaks = 2^seq(-4, 4, 2)) +
geom_hline(yintercept = 1) +
labs(x = "Intensity", y = "Fold Change, treated vs control") +
facet_grid(rows = vars(treated_sample), cols = vars(control_sample))
样品4的丰度变化现在显示得更清楚了。 这不是一个常见的绘制数据的方法,所以可能需要向你的同事解释一下,但在我看来是值得努力的。